Per le sue caratteristiche (dimensione del genoma, rapidità nella crescita e nello sviluppo, semplicità nella propagazione e coltivazione) il pioppo è utilizzato come modello per gli studi di genetica e genomica, messo a confronto, attraverso analisi, con altre specie. L’individuazione dei geni che regolano determinati caratteri e lo sviluppo dei relativi marcatori molecolari permettono di implementare i programmi di breeding mediante la selezione assistita da marcatori molecolari, che consente la selezione precoce dei caratteri ritenuti interessanti. Scopriamo insieme le ricerche dei centri Foreste e Legno e Genomica e Bioinformatica
Il pioppo come pianta modello
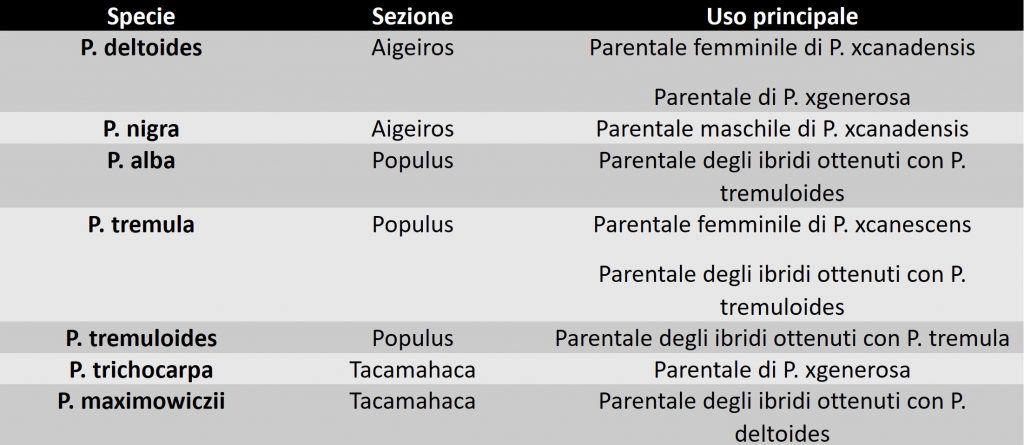
Il genere Populus include 30 specie tassonomicamente classificate nelle sei sezioni Populus, Tacamahaca, Leucoides, Turanga, Abaso e Aigeiros (Eckenwalder, 1996). Le specie Aigeiros, Populus e Tacamahaca rappresentano quelle utilizzate per le attività di breeding e per lo sviluppo di cloni commerciali.
Nell’emisfero nord il pioppo ricopre importanti ruoli economici, in particolare per l’industria cartiera, la produzione di compensato, la decontaminazione dei suoli dai metalli pesanti, il recupero di ecosistemi fragili, come quelli esposti a desertificazione, e il ripristino dei paesaggi forestali (Bergante e Facciotto, 2015).
Grazie alla piccola dimensione del genoma di circa 500 Mb, suddivise in 19 cromosomi (Tuskan et al., 2006), alla crescita e sviluppo rapidi e ai semplici metodi di propagazione e coltivazione, il pioppo è diventato la specie modello per gli studi di genetica e genomica (Carletti et al., 2016).
Il sequenziamento del genoma di P. trichocarpa (https://mycocosm.jgi.doe.gov/Poptr1_1/Poptr1_1.home.html; Tuskan et al., 2006) è stato fondamentale per gli studi di genetica delle piante arboree, essendo stato il primo genoma di una specie forestale ad essere pubblicato. Successivamente a questo primo sequenziamento, sono state rilasciate altre sequenze genomiche di riferimento: P. deltoides (Bai et al., 2021), P. pruinosa (Yang et al., 2017a), P. euphratica (Ma et al., 2013; Zhang et al., 2020), P. tremula e P. tremuloides (Lin et al., 2018), P. alba (Liu et al., 2019; Ma et al., 2019), P. simonii (Wu et al., 2020) e l’ibrido P. alba x P. tremula varietà glandulosa 84K (Qiu et al., 2019; Huang et al., 2021).
Tali risorse genetiche sono state basilari per le analisi della diversità genetica presente in pioppo e per la costruzione di un pan-genoma rappresentativo delle varianti genetiche strutturali presenti in questa specie (Pinosio et al., 2016). Inoltre, l’assemblaggio dei genomi di pioppo ha aperto la strada allo sviluppo di migliaia di marcatori molecolari,basati su mutazioni di singole basi del DNA (Single Nucleotide Polymorphism – SNP) e distribuiti su tutto il genoma, i quali consentono di sviluppare dei sistemi automatizzati e a basso costo per la genotipizzazione (ossia la definizione delle differenze nel corredo genetico di invidui tramite l’esame della sequenza del DNA) simultanea di centinaia/migliaia di piante diverse
Il progetto europeo B4EST (Adaptive BREEDING for Better FORESTs)
Nell’ambito del progetto europeo B4EST (Adaptive BREEDING for Better FORESTs; https://b4est.eu/), il CREA in collaborazione con altri centri di ricerca europei, ha contribuito a sviluppare 13400 marcatori molecolari SNP, distribuiti su tutto il genoma di pioppo, che sono stati inseriti in un sistema commerciale di genotipizzazione di quattro specie forestali, denominato 4TREE array e caratterizzato da un supporto solido, al quale sono associate migliaia di microscopici segmenti di DNA (sonde), ognuno corrispondente a uno specifico SNP, i quali, in seguito a ibridazione con il DNA di un inviduo, permettono di definire le varianti di sequenza ad ogni marcatore. Il 4TREE array è stato utilizzato per genotipizzare due popolazioni di pioppo costituite da centinaia di piante derivanti dall’incrocio tra individui resistenti all’afide lanigero (Woolly poplar aphid – WPA) con individui suscettibili ad esso. E’ stata inoltre valutata la resistenza/suscettibilità all’afide di ognuno degli individui delle popolazioni e i dati sono stati associati ai dati genetici registrati in precedenza. Tale analisi ha portato alla definizione di regioni del genoma di pioppo implicate nella resistenza a WPA.

La selezione genomica in pioppo
L’individuazione dei geni/loci che regolano determinati caratteri e lo sviluppo dei marcatori molecolari associati ad essi, permettono di implementare i programmi di breeding mediante la selezione assistita da marcatori molecolari, che consente la selezione precoce per caratteri fenotipici di interesse, evitando le costose fenotipizzazioni (ndr ossia l’analisi visiva delle caratteristiche morfologiche e funzionali), che richiedono molto tempo, spazio e forza lavoro. Tuttavia, il successo alla base di un ibrido di pioppo è dovuto alla combinazione di numerosi geni diversi, ciascuno dei quali contribuisce con un piccolo o piccolissimo effetto alla performance complessiva della pianta. Esempi di caratteri di rilevanza del pioppo, controllati da decine o centinaia di geni, sono la qualità del legno, la velocità di accrescimento e la resistenza ad avversità biotiche ed abiotiche.
Per valutare l’effetto complessivo di tutti questi geni sulle caratteristiche morfologiche e funzionali oggetto di studio durante il processo di selezione, è possibile utilizzare la predizione genomica (Genomic Prediction – GP). La GP usa modelli matematici per combinare dati fenotipici e dati relativi ai marcatori molecolari per stimare il valore genetico degli individui. In questo modo è possibile predire la manifestazione di un carattere di un individuo analizzando solo il suo DNA. I modelli matematici vengono calcolati integrando i dati genetici e le caratteristiche morfo-funzionali registrate per una popolazione di riferimento (training population) costituita da poche centinaia di individui. Successivamente, essi vengono utilizzati per stimare il fenotipo di qualsiasi altro individuo, per esempio piante di pioppo da selezionare, a partire soltanto da dati genetici.

Nel pioppo, così come in altre specie arboree, l’espressione dei caratteri di interesse, quali quelli legati alla qualità del legno, avviene nelle piante adulte le quali raggiungono la maturità in circa 7-8 anni. In piante con un lungo ciclo di crescita come questa specie, la GP è uno strumento che permette di selezionare le piante giovani che ancora non esprimono i caratteri di interesse e, quindi, di ridurre al minimo il numero di piante da fenotipizzare nelle prove di campo.
Calcolando l’effetto dei singoli marcatori sui caratteri di interesse, rispetto al tradizionale miglioramento genetico del pioppo basato sulla selezione fenotipica, la GP permette di stimare in anticipo il valore genetico degli individui, riducendo la necessità di estese fenotipizzazioni in campo. Inoltre, diversi studi hanno dimostrato che la GP permette di realizzare un uguale o maggiore progresso genetico per unità di tempo, ad un costo inferiore rispetto alla selezione fenotipica.
Fino a pochi anni fa, il numero limitato di marcatori molecolari disponibile per il pioppo ha ostacolato l’applicazione della GP in questa specie. Le nuove risorse genomiche, come il 4TREE array, hanno contribuito ad implementare la GP nel pioppo e di conseguenza a rendere più efficiente il miglioramento genetico di questa specie.
Il CREA, sfruttando le conoscenze generate nell’ambito del progetto B4EST, ha deciso di utilizzare la GP nei propri programmi di miglioramento genetico del pioppo, in particolare per selezionare piante più resistenti all’afide lanigero e con una migliore qualità del legno.
La cisgenesi e il genome editing in pioppo

La disponibilità di un genoma sequenziato ha aperto la strada anche alle tecniche di evoluzione assistita (TEA) quali cisgenesi e genome editing (GE), con cui è possibile modificare in modo mirato cloni di pioppo di pregio, inserendo caratteri utili per l’adattamento alle condizioni ambientali e la produttività, senza alterare il background genetico e le caratteristiche agronomiche positive.
La cisgenesi consente il trasferimento di geni tra cloni o anche specie diverse, purché sessualmente compatibili, senza l’introduzione di DNA estraneo, realizzando in un tempo relativamente breve un risultato che sarebbe conseguibile attraverso una serie di incroci successivi in programmi pluriennali di miglioramento genetico classico. Dal punto di vista tecnico la cisgenesi si basa sull’utilizzo di vettori di trasformazione marker-free, che consentono di eliminare i marcatori molecolari necessari alla selezione delle piante modificate. Ad esempio, è stato possibile incrementare il tasso di accrescimento, inserendo in cloni di pioppo una copia supplementare del gene GA20ox7 che codifica per un enzima responsabile della biosintesi delle gibberelline, ormoni vegetali che stimolano la crescita (Han et al., 2011): si è dimostrato così che l’introduzione di una copia supplementare di un gene già presente nel genoma ricevente è sufficiente per ottenere un effetto fenotipico. Effetti fenotipici ancora maggiori sono potenzialmente conseguibili con l’utilizzo di alleli superiori dei geni di interesse, la cui individuazione è oggi in corso grazie all’applicazione di tecniche di mappaggio genico avanzate.
Da tempo sono in corso al CREA programmi di ricerca per l’utilizzo di vettori di trasformazione marker-free (Zelasco et al., 2007). Recentemente, in prove di cisgenesi condotte nell’ambito del progetto Biotech (https://www.crea.gov.it/en/-/biotech) sono state prodotte una serie di linee di pioppo nero (P. nigra clone Jean Pourtet) modificate per la produzione di gibberelline, con l’obiettivo di incrementare la crescita e migliorare la resa in biomassa per la produzione di biocarburanti. Per quest’ultimo obiettivo è stato utilizzato il gene DUF266, che codifica per un enzima coinvolto nella biosintesi della cellulosa (Yang et al., 2017b).

La richiesta di biomasse di seconda generazione è prevista in forte aumento nei prossimi anni per soddisfare il fabbisogno di biocarburanti in vista dell’obiettivo di ridurre a zero le emissioni nette di anidride carbonica per il 2050 (IEA, 2022). Piante per la produzione di biomasse fermentabili come il pioppo presentano il vantaggio di poter essere coltivate in modo poco intensivo, anche in terreni marginali, senza sottrarre suolo agrario alle colture alimentari. Il maggiore svantaggio, per contro, è rappresentato dall’impatto economico ed ambientale dei pretrattamenti chimico-fisici necessari per la separazione della lignina dalla massa fermentabile. L’approccio cisgenico che prevede l’inserimento del gene DUF266 risponde a questa limitazione, perché è stato visto che la sovraespressione di questo gene consente di ottenere biomasse lignocellulosiche, che necessitano di pretrattamenti meno onerosi (Yang et al., 2017b).

Il CREA ha avviato, sempre nell’ambito del progetto Biotech, esperimenti per la modificazione della composizione del legno anche attraverso GE. Questa tecnica, che consente di inattivare o modificare in modo mirato specifici geni, è stata utilizzata in pioppo per modificare caratteri relativi a resistenza a stress biotici e qualità del legno (Bewg et al., 2018; Anders et al., 2023). Presso il Centro Foreste e Legno (CREA-FL) sono state ottenute numerose linee di pioppo bianco (P. alba clone Villafranca) modificate con GE. A tale scopo sono stati scelti i geni CAD (Cinnamil-alcol deidrogenasi) e C3H (p-cumarato 3-idrossilasi), che codificano per enzimi della via di biosintesi della lignina; inattivando tali geni si sono ottenute piante più adatte alla produzione di biomassa lignocellulosica da avviare alla produzione di biocarburanti. Attualmente, queste linee sono oggetto di studio per la verifica della presenza di modifiche a carico del DNA, con effetto sulla composizione e la quantità della lignina.
Bibliografia
Anders, C.; Hoengenaert, L.; Boerjan, W. (2023) Accelerating wood domestication in forest trees through genome editing: Advances and prospects. Curr. Op. Plant Biol. 71:102329. Doi: 10.1016/j.pbi.2022.102329.
Bai, S.; Wu, H.; Zhang, J.; Pan, Z.; Zhao, W.; Li, Z.; Tong, C. (2021) Genome assembly of Salicaceae Populus deltoides (eastern cottonwood) i-69 based on nanopore sequencing and hi-c technologies. J. Hered. 112:303-310. Doi: 10.1093/jhered/esab010.
Bergante, S.; Facciotto, G. (2015) Yields of poplars SRC and VSRC grown with different fertilization and irrigation management. Proceedings of the 23th European Biomass Conference and Exhibition, Vienna, Austria, 1-4 June 2015; pp. 1-4.
Bewg, W.P.; Ci, D.; Tsai, C.J. (2018) Genome editing in trees: from multiple repair pathways to long-term stability. Front. Plant Sci. 9:1732. Doi: 10.3389/fpls.2018.01732/full.
Biselli, C.; Vietto, L.; Rosso, L.; Cattivelli, L.; Nervo, G.; Fricano, A. (2022) Advance Breeding for Biotic Stress Resistance in Poplar. Plants 11:2032. Doi:10.3390/plants11152032.
Carletti, G., Carra, A.; Allegro, G.; Vietto, L.; Desiderio, F.; Bagnaresi, P.; Gianinetti, A.; Cattivelli, L.; Valè, G.; Nervo, G. (2016) QTLs for Woolly Poplar aphid (Phloeomyzus passerinii L.) resistance detected in an inter-specific Populus deltoides x P. nigra mapping population. PLoS ONE, 11(3). Doi: 10.1371/journal.pone.0152569.
Faivre-Rampant, P.; Zaina, G.; Jorge, V.; Giacomello, S.; Segura, V.; Scalabrin, S.; Guerin, V.; De Paoli, E.; Aluome, C.; Viger, M.; et al. (2016) New resources for genetic studies in Populus nigra: Genome-wide SNP discovery and development of a 12k Infinium array. Mol. Ecol. Resour. 16:1023–1036. Doi: 10.1111/1755-0998.12513.
Geraldes, A.; DiFazio, S.P.; Slavov, G.T.; Ranjan, P.; Muchero, W.; Hannemann, J.; Gunter, L.E.; Wymore, A.M.; Grassa, C.J.; Farzaneh, N.; et al. (2013) A 34K SNP genotyping array for P. trichocarpa: Design, application to the study of natural populations and transferability to other Populus species. Mol. Ecol. Resour. 13:306-323. Doi: 10.1111/1755-0998.12056.
Eckenwalder, J. E. (1996) ‘Systematics and evolution of Populus’, in Biology of Populus and its Implications for Management and Conservation Part I.
Han, K.M.; Dharmawardhana, P.; Arias, R.S.; Ma, C.; Busov, V.; Strauss, S.H. (2011) Gibberellin‐associated cisgenes modify growth, stature and wood properties in Populus. Plant Biotechnol. J. 9(2):162-178. Doi: 10.1111/j.1467-7652.2010.00537.x.
Huang, X.; Chen, S.; Peng, X.; Bae, E.K.; Dai, X.; Liu, G.; Qu, G.; Ko, J.H.; Lee, H.; Chen, S.; et al. (2021) An improved draft genome sequence of hybrid Populus alba x Populus glandulosa. J. For. Res. 32:1663-1672. Doi: 10.1007/s11676-020-01235-2.
IEA, International Energy Agency. Renewables 2022: Analysis and forecast to 2027.
Lin, Y.C.; Wang, J.; Delhomme, N.; Schiffthaler, B.; Sundstrom, G.; Zuccolo, A.; Nystedt, B.; Hvidsten, T.R.; de la Torre, A.; Cossu, R.M.; et al. (2018) Functional and evolutionary genomic inferences in Populus through genome and population sequencing of American and European aspen. Proc. Natl. Acad. Sci. USA 115:E10970-E10978. Doi: 10.1073/pnas.1801437115.
Liu, Y.J., Wang, X.R.; Zeng, Q.Y. (2019) De novo assembly of white poplar genome and genetic diversity of white poplar population in Irtysh River basin in China. Sci. China Life Sci. 62:609-618. Doi: 10.1007/s11427-018-9455-2.
Ma, T.; Wang, J.; Zhou, G.; Yue, Z.; Hu, Q.; Che, Y.; Liu, B.; Qiu, Q.; Wnag, Z.; Zhang, J.; et al. (2013) Genomic insights into salt adaptation in a desert poplar. Nat. Commun. 4:2797. Doi: 10.1038/ncomms3797.
Ma, J.; Wa, D.; Duan, B.; Bai, X.; Bai, Q.; Chen, N.; Ma, T. (2019) Genome sequence and genetic transformation of a widely distributed and cultivated poplar. Plant Biotechnol. J. 17:451-460. Doi: 10.1111/pbi.12989.
Pinosio, S.; Giacomello, S.; Faivre-Rampant, P.; Taylor, G.; Jorge, V.; Le Paslier, M.C.; Zaina, G.; Bastien, C.; Cattonaro, F.; Marroni, F.; et al. (2016) Characterization of the Poplar Pan-Genome by Genome-Wide Identification of Structural Variation. Mol. Biol. Evol. 33:2706-2719. Doi: 10.1093/molbev/msw161.
Qiu, D.; Bai, S.; Ma, J.; Zhang, L.; Shao, F.; Zhang, K.; Yang, Y.; Sun, T.; Huang, J.; Zhou, Y.; et al. (2019) The genome of Populus alba x Populus tremula var. glandulosa clone 84K. DNA Res. 26:423-431. Doi: 10.1093/dnares/dsz020.
Scaglione, D.; Pinosio, S.; Marroni, F.; Centa, E.; Di Fornasiero, A.; Magris, G.; Scalabrin, S.; Cattonaro, F.; Taylor, G.; Morgante, M. (2019) Single primer enrichment technology as a tool for massive genotyping: A benchmark on black poplar and maize. Ann. Bot. 124:543-551. Doi: 10.1093/aob/mcz054.
Schilling, M.P.; Wolf, P.G.; Duffy, A.M.; Rai, H.S.; Rowe, C.A.; Richardson, B.A.; Mock, K.E. (2014) Genotyping-by-sequencing for Populus population genomics: An assessment of genome sampling patterns and filtering approaches. PLoS ONE 9:e95292. Doi: 10.1371/journal.pone.0095292.
Tuskan, G.A.; DiFazio, S.; Jansson, S.; Bohlmann, J.; Grigoriev, L.; Hellsten, U.; Putnam, N.; Ralph, S.; Rombauts, S.; Salamov, A.; et al. (2006) The Genome of Black Cottonwood, Populus trichocarpa (Torr. & Gray). Science, 313(5793): 1596–1604. Available at: http://science.sciencemag.org/content/313/5793/1596.abstract.
Wu, H.; Yao, D.; Chen, Y.; Yang, W.; Zhao, W.; Gao, H.; Tong, C. (2020) De novo genome assembly of Populus simonii further supports that Populus simonii and Populus trichocarpa belong to different sections. G3 Genes Genom. Genet. 10:455-466. Doi: 10.1534/g3.119.400913.
Yang, W.; Wang, K.; Zhang, J.; Ma, J.; Liu, J.; Ma, T. (2017a) The draft genome sequence of a desert tree Populus pruinosa. GigaScience 6:2797. Doi: 10.1093/gigascience/gix075.
Yang, Y.; Yoo, C. G.; Guo, H. B.; Rottmann, W.; Winkeler, K. A.; Collins, C. M.; Gunter, L.E.; Jawdy, S.S.; Yang, X.; Guo, H.; Pu, Y.; Ragauskas, A.J.; Tuskan, G.A.; Chen, J. G. (2017b). Overexpression of a Domain of Unknown Function 266-containing protein results in high cellulose content, reduced recalcitrance, and enhanced plant growth in the bioenergy crop Populus. Biotechnol. Biofuels, 10, 1-13. DOI https://doi.org/10.1186/s13068-017-0760-x.
Zelasco, S.; Ressegotti, V.; Confalonieri, M.; Carbonera, D.; Calligari, P.; Bonadei, M.; … & Balestrazzi, A. (2007) Evaluation of MAT-vector system in white poplar (Populus alba L.) and production of ipt marker-free transgenic plants by ‘single-step transformation’. Plant cell tissue organ cult. 91:61-72. Doi: 10.1007/s11240-007-9278-4.
Zhang, Z.; Chen, Y.; Zhang, J.; Ma, X.; Li, Y.; Li, M.; Wang, D.; Kang, M.; Wu, H.; Yang, Y.; et al. (2020) Improved genome assembly provides new insights into genome evolution in a desert poplar (Populus euphratica). Mol. Ecol. Resour. 20:781-794. Doi: 10.1111/1755-0998.13142.

Ricercatore CREA Centro Foreste e Legno
Lavora nel campo della genomica e delle biotecnologie applicate al pioppo. In precedenza si è occupato di microRNA della vite e del pomodoro.
#lafrase “Ben detto”, rispose Candido, “ma dobbiamo coltivare il nostro orto.” (Voltaire)

Ricercatrice CREA Centro Viticoltura ed Enologia
Ambito di Ricerca: Genetica, Genomica, Interazione pianta-patogeno
#lafrase La potenza genetica è la forza più dirompente che esista (Jurassic Park)

CREA Centro Genomica e Bioinformatica
Studia la struttura e la funzione dei genomi e dei modelli per la predizione genomica
#lafrase Diffondere e promuovere la conoscenza e l’applicazione del metodo scientifico significa migliorare la nostra società.

Dirigente di ricerca, CREA Centro Foreste e Legno
già direttore dell’Istituto di Sperimentazione per la pioppicoltura di Casale Monferrato, membro dell’Osservatorio Nazionale del Pioppo presso il Ministero dell’agricoltura, della sovranità alimentare e delle foreste (Masaf) e del Comitato esecutivo dell’ International Commission on Poplars and Other Fast-Growing Trees Sustaining People and the Environment (IPC).
#lafrase Gli alberi sono poesie che la terra scrive in cielo (Khalil Gibran)




[…] 45,000 to 50,000 hectares of poplar, mainly in the Po River plain on agricultural land. The wood produced is used for various industrial and energy purposes, with a predominant demand in producing plywood […]